Pytania i odpowiedzi
Oczyszczanie i metody badań makrocząsteczek życia
Zebrane pytania i odpowiedzi do zestawu.
Ilość pytań: 136
Rozwiązywany: 5132 razy
Pytanie 101
Reakcja PCR w której przeprowadzamy amplifikację więcej niż jednego odcinka DNA nazywa się:
d. multipleks PCR
Pytanie 102
Co jest metodą do RNA?
c. RT-PCR
Pytanie 103
RT-qPCR to:
b. Reakcja PCR w czasie rzeczywistym (Real Time)
Pytanie 104
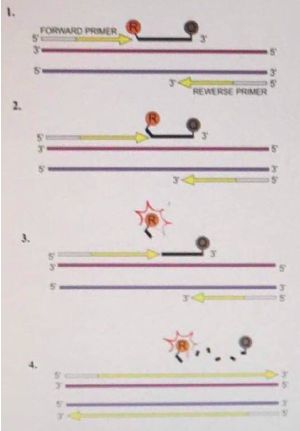
Metodę RT-qPCR z wykorzystaniem jakich barwników/sond przedstawia powyższa rycina? Opis do ryciny: 1.
polimeryzacja – reporter (R) i wygaszacz (Q) są związane do oligonukleotydowej sondy; 2. przemieszczenie się nici –
wygaszacz nadal pochłania sygnał emitowany przez reporter; 3. rozłam – wygaszacz odłącza się od sondy i oddala od
reportera – sygnał staje się widoczny; 4. zakończenie polimeryzacji – odłączony reporter nadal emituje sygnał
fluorescencji.
a. SYBR Green 1
Pytanie 105
Mikromacierze RNA dostarczają nam informacji o:
d. profilach jakościowej i ilościowej ekspresji genów
Pytanie 106
Wskaż zdanie FAŁSZYWE. Mikromacierze DNA:
d. metoda, w której stosowane są dwuniciowe sondy w postaci DNA o znanej sekwencji, produktów PCR lub oligonukleotydów
Pytanie 107
Wykrywanie ekspresji genów, przy pomocy mikromacierzy jest możliwe dzięki temu, że:
d. wszystkie z wymienionych
Pytanie 108
Mikromacierze DNA to technologia, która umożliwia badanie i rozwiązywanie problemów biomedycznych. Które były
kiedyś uważane za nierozwiązywalne dlatego że:
d. Wszystkie z wymienionych
Pytanie 109
W metodzie mikromacierzy:
a. do sond umieszczonych na stałym podłożu dodaje się wyznakowany DNA badany b. do sond umieszczonych na stałym podłożu dodaje się niewyznakowany DNA badany c. do badanego wyznakowanego DNA umieszczonego na stałym podłożu dodaje się wiele sond d. do badanego niewyznakowanego DNA umieszczonego na stałym podłożu dodaje się wiele sond
Pytanie 110
Wskaż zdanie FAŁSZYWE dotyczące mikromacierzy:
b. Niska specyficzność wobec genów
Pytanie 111
Mikromacierze SNP są ‘lepsze” od porównawczej hybrydyzacji genomowej, gdyż:
c. nie można porównać tych dwóch technik, gdyż mikromacierze SNP służą do analizy pojedynczych genów/nukleotydów, a porównawcza hybrydyzacja genomowa do analizy całych genomów
Pytanie 112
Metoda, w której stosuję sie sondę oligonukleotydową do ustalenia, która z dwóch alternatywnych sekwencji
występuje w danej cząsteczce to:
c. Hybrydyzacja allelospecyficzna
Pytanie 113
Technika FISH:
d. Stanowi modyfikację metody hybrydyzacji in situ
Pytanie 114
Podaj nazwę techniki, w której izolowany DNA z jądra komórkowego rozciąga się i utrwala na szkiełku
podstawowym:
c. Fiber FISH
Pytanie 115
Metoda biologii molekularnej wykorzystująca różnicę w ilości i rodzaju miejsc restrykcyjnych w badanym fragmencie
materiały genetycznego to technika:
b. RFLP
Pytanie 116
Rolą enzymów restrykcyjnych w klonowaniu DNA jest:
b. wytworzenie lepkich końców wokół genu do sklonowania i w plazmidzie, w celu wstawienia genu do plazmidu
Pytanie 117
W reakcja hybrydyzacji z DNA sonda molekularna:
a. powinna być wyznakowana w celu zidentyfikowania powstałych dwuniciowych kompleksów
Pytanie 118
Lepkie końce powstają w wyniku cięcia nici DNA:
c. Enzymem restrykcyjnym
Pytanie 119
Enzym restrykcyjny:
d. rozpoznaje i przecina tylko określone sekwencje nukleotydowe w cząsteczce DNA
Pytanie 120
Hybrydyzacja Southern Blot:
b. Wykrywa mutacje punktowe


