Nauka
Oczyszczanie i metody badań makrocząsteczek życia
Wyświetlane są wszystkie pytania.
Pytanie 65
Wskaż nieprawidłowe stwierdzenie dotyczące sekwencjonowania cyklicznego:
b. do reakcji amplifikacji używany jest jeden starter
c. produkt reakcji przyrasta wykładniczo
a. ze względu na obecność dideoksynuklotydów dochodzi do terminacji łańcucha
d. materiałem wyjściowym jest dwuniciowy DNA
Pytanie 66
Dideoksynukleotydy to:
a. zdenaturowane nukleotydy powstające w trakcie oczyszczania produktu PCR przed sekwencjonowaniem
b. trifosforany dideoksynukleozydów, które są analogami nukleotydów uniemożliwiającymi przyłączenie do syntetyzowanego łańcucha DNA następnego nukleotydu
c. trifisforany dideoksynukleozydów, które są znakowanymi starterami pozwalającymi na specyficzne namnażanie fragmentów PCR do sekwencjonowania
d. nukleotydy podlegające modyfikacji w trakcie reakcji sekwencjonowania
Pytanie 67
Sekwencjonowanie metodą chemicznej degradacji to inna nazwa:
d. metody Maxama-Gilberta
b. pirosekwencjonowania
a. sekwencjonowania cyklicznego
c. metody Sangera
Pytanie 68
“Jest najstarszą metodą sekwencjonowania klasycznego opartą na chemicznej degradacji łańcucha DNA. Składa się z
3 etapów : chemicznej modyfikacji zasady azotowej, usunięciu tej zasady i przecięciu nici DNA w miejscu AP. Matrycę
do sekwencjonowania w tej metodzie może stanowić jedno- lub dwuniciowy DNA, wyznakowany radioaktywnie na
końcu 5’”. Jaka to metoda:
c. metoda Maxama-Gilberta
b. metoda Sangera
a. sekwencjonowanie masowe
d. pirosekwencjonowanie
Pytanie 69
Jak nazywa się metoda sekwencjonowania peptydów polegająca na kolejnym odrywaniu oznakowanych
aminokwasów z N-końca cząsteczki peptydu?
b. sekwencjonowanie Sangera
a. sekwencjonowanie równoległe
c. degradacja Edmana
d. degradacja równoległa
Pytanie 70
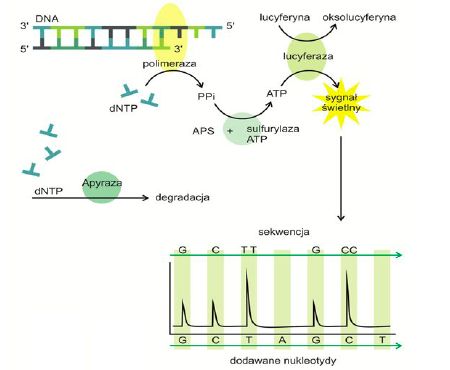
Jaką metodę przedstawia poniższa rycina?
a. Pirosekwencjowanie, które należy do NGS
c. Sekwencjonowanie nanoporowe, które jest sekwencjonowaniem klasycznym
d. Metoda Sangera, które jest sekwencjonowaniem klasycznym
b. Metoda Solexa, które należy do NGS
Pytanie 71
W przypadku sekwencjonowania DNA metodą Sanger’a, kolejność nukleotydów w badanym DNA ustalimy poprzez
c. analizę intensywności fluorescencji hybrydyzowanej sondy
b. analizę widma absorpcyjnego
a. analizę prążków reakcji PCR sekwencyjnego
d. analizę produktów cięcia enzymami restrykcyjnymi
Pytanie 72
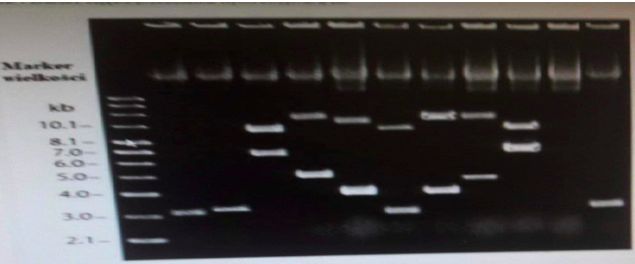
Poniższe zdjęcie przedstawia wynik otrzymany po:
b. Real Time PCR
d. Hybrydyzacji FISH
a. sekwencjonowaniu Sangera
c. Elektroforezie